在研究一个基因时,常常会需要了解这个基因的(核苷酸或氨基酸)序列在物种间的保守性。下面提供一种查询基因同源情况、序列比对及可视化的方法。以基因 SF3B1 为例:
基因在物种间的保守性
如果是well-known的基因,保守性大概了解,可以自行判断是使用核苷酸序列(非常保守时使用)还是氨基酸序列(不怎么保守时使用)。
如果未知,可以通过以下途径知晓:
- blast/blastp,可以直接用human里面该基因的序列进行blast/blastp比对,查看比对到有哪些物种。
- OrthoDB. 在OrthoDB用基因名查询。
获取物种进化关系
如果你想显示有哪些物种,可以使用TimeTree绘制指定物种间的进化树。
多条序列比较
确定要比较的物种,获取这些物种SF3B1的序列,用 snapGene 进行多序列比对。
多比对的结果 export为fasta格式。
!!!如果比对结果中,个别地方有碱基的错位,可手动编辑fasta文件,进行修改。
比对结果可视化
下面提供一个在线的简单可视化工具:碱基顺序比对工具
将比对结果fasta文件输入后,submit,出现的结果,可点击鼠标右键,直接打印输出为PDF文件。或者复制存储为其他类型文件。

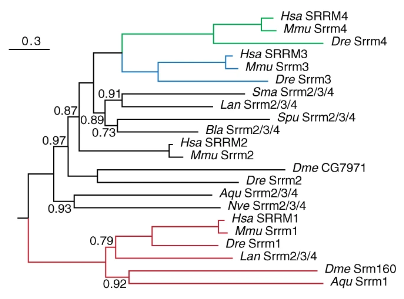
序列进化树
序列进化也是一个常规的需求,也不乏成熟的方法。
参考1:A novel protein domain in an ancestral splicing factor drove the evolution of neural microexons
method: Search for Srrm4 orthologues and generation of multi-species alignment